Människans genom: definition och översikt av arvsmassa, HGP och ENCODE
Upptäck människans genom: definition, arvsmassa, Human Genome Project (HGP) och ENCODE — hur DNA-sekvenser och genreglering formar biologi och medicin.
Den mänskliga arvsmassan är lagrad på 23 kromosompar i cellkärnan och i det lilla mitokondriella DNA:t. Haploidgenomet innehåller ungefär 3,1 miljarder baspar. Av dessa utgörs en relativt liten del av gener som kodar för proteiner (≈20 000 proteinkodande gener), medan stora delar är icke‑kodande DNA som kan ha regulatoriska funktioner, vara involverat i kromosomstruktur eller vara kvarlevor av gamla transponibla element. Utöver proteinkodande gener finns många icke‑kodande RNA (t.ex. tRNA, rRNA och långa icke‑kodande RNA) som fyller viktiga biologiska roller. Vetenskapen har kartlagt många av DNA-sekvenser på våra kromosomer och förstår i allt större utsträckning hur olika delar fungerar, men mycket återstår att lära för att fullt ut kunna tolka och använda denna information i medicin och biologi.
Human Genome Project (HGP) — referenssekvensen
Human Genome Project (HGP) var ett internationellt samarbete som pågick under 1990‑talet och början av 2000‑talet med målet att bestämma en referenssekvens för människans genom. Projektet producerade en referenssekvens som används över hela världen inom biologi och medicin. Nature publicerade rapporten från det offentligt finansierade projektet och Science publicerade Celeras artikel. Dessa artiklar beskrev hur utkastet till sekvens producerades och gav en analys av sekvensen.
Det första utkastet publicerades 2001 och innehöll stora framsteg i förståelsen av geninnehåll och kromosomstruktur. Förbättrade utkast tillkännagavs 2003 och 2005, som fyllde på till ≈92 % av sekvensen vid den tidpunkten. Sedan dess har tekniska framsteg — särskilt nästa generations sekvensering och senare långläsningsmetoder (t.ex. PacBio och Oxford Nanopore) — gjort det möjligt att slutföra och förbättra referenssekvenserna ytterligare. Moderna referenssamlingar (t.ex. GRCh38) och nyare projekt som Telomere‑to‑Telomere (T2T‑CHM13) har arbetat för att täcka tidigare outredda regioner, inklusive repetitiva delar och telomerer.
ENCODE — att förstå genreglering
Det senaste storskaliga projektet ENCODE (ENCyclopedia Of DNA Elements) startade för att systematiskt kartlägga vilka delar av genomet som är biologiskt aktiva och hur gener styrs. ENCODE använder många typer av experimentella metoder (t.ex. RNA‑seq för transkript, ChIP‑seq för bindning av transkriptionsfaktorer och histonmodifieringar, DNase‑seq/ATAC‑seq för öppen kromatin) för att identifiera promotorer, enhancers, transkriptionsfaktorbindningsställen och andra regulatoriska element.
Resultaten från ENCODE visade att en stor andel av genomet visar någon form av biokemisk aktivitet i ett eller flera celltyper. Detta har lett till diskussioner om hur man ska definiera "funktion" — om biokemisk aktivitet per automatik innebär biologisk funktion eller om bevis för selektion och fysiologisk effekt behövs. Trots denna debatt har ENCODE bidragit kraftigt till vår förståelse av genreglering och gett verktyg och data som används för att tolka genetiska varianter, särskilt de som ligger utanför proteinkodande regioner.
Användningar, begränsningar och etiska aspekter
- Medicinsk tillämpning: Referenssekvenser och data från HGP/ENCODE ligger till grund för diagnostik (t.ex. panel‑ och exomsekvensering), cancergenomik, farmakogenetik och identifiering av sjukdomsorsakande varianter.
- Forskning: Genomsdata används för att kartlägga gen‑funktion, evolutionära studier, populationgenetik och för att utveckla nya metoder för behandling och biomarkörer.
- Tekniska begränsningar: Tolkningsproblem kvarstår — många upptäckta varianter har oklar klinisk betydelse, och repetitiva eller strukturella regioner är svåra att sekvensera och sammanfoga. Heterogenitet mellan individer kräver också större och mer diversifierade referensdatabaser (pangenom‑initiativ).
- Etik och samhälle: Genomisk information väcker frågor om integritet, samtycke, genetisk diskriminering och rättvis tillgång till ny teknik. Ansvarsfull datahantering och tydlig kommunikation med patienter och deltagare är nödvändiga.
Framtiden
Framtida arbete fokuserar på att få mer fullständiga och representativa referenser (inklusive pangenom), förbättra tolkningen av icke‑kodande varianter, integrera genomsdata med andra "omik"‑lager (proteomik, epigenomik, metabolomik) och utveckla precisionsmedicin som tar hänsyn till genetisk variation i olika befolkningsgrupper. Fortsatt teknikutveckling och internationellt samarbete kommer att vara avgörande för att skörda den fulla nyttan av kunskapen om människans genom.
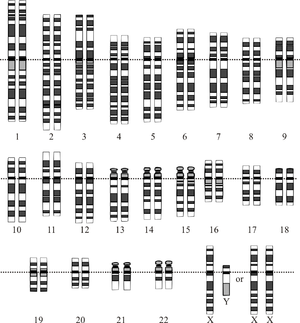
Grafisk representation av den idealiserade mänskliga karyotypen, som visar hur genomet är indelat i kromosomer. Denna ritning visar både den kvinnliga (XX) och manliga (XY) versionen av det 23:e kromosomparet.
DNA och proteiner
Den mänskliga arvsmassan innehåller drygt 20 000 proteinkodande gener, vilket är betydligt färre än vad man hade förväntat sig. Faktum är att endast cirka 1,5 % av genomet kodar för proteiner, medan resten består av icke-kodande RNA-gener, regleringssekvenser och introner.
En enda gen kan dock producera en mängd olika proteiner genom RNA-splicing. En viss Drosophila-gen (DSCAM) kan splicas alternativt till 38 000 olika mRNA:er. Varje mRNA kodar för en annan peptidkedja. Därför är antalet producerade proteiner långt större än antalet kodande gener.
Med RNA-splicing och förändringar efter RNA-translation kan det totala antalet unika mänskliga proteiner vara i flera miljoner.
Idén att det mesta DNA är värdelöst skräp är felaktig. Minst 80 % av arvsmassan har bestämda funktioner.
Skillnader mellan människor och schimpanser
Det djur som lever nu och som står människan närmast är schimpansen. 98,4 % av DNA:t är detsamma mellan människor och schimpanser. Detta gäller dock endast för polymorfismer i enstaka nukleotider, det vill säga förändringar i endast enstaka baspar. Den fullständiga bilden är ganska annorlunda.
Utkastet till sekvensen av schimpansens gemensamma genom publicerades 2005. Den visade att de regioner som är tillräckligt lika för att kunna anpassas till varandra utgör 2400 miljoner av människans 3164,7 miljoner baser, dvs. 75,8 % av genomet.
Dessa 75,8 % av människans genom skiljer sig 1,23 % från schimpansens genom när det gäller polymorfismer i enskilda nukleotider (SNPs - förändringar av enskilda DNA-"bokstäver" i genomet). En annan typ av skillnader, s.k. indels (inskjutningar/utplåningar), står för ytterligare ~3 % av skillnaderna mellan de aligerbara sekvenserna. Dessutom ger variationen i antalet kopior av stora segment (> 20 kb) av liknande DNA-sekvenser ytterligare 2,7 % skillnad mellan de två arterna. Den totala likheten mellan genomerna kan därför vara så låg som ca 70 %.
Relaterade sidor
Frågor och svar
F: Var lagras det mänskliga genomet?
S: Det mänskliga genomet finns lagrat på 23 kromosompar i cellkärnan och i det lilla mitokondriella DNA:t.
F: Vad vet man idag om DNA-sekvenserna på våra kromosomer?
S: Mycket är nu känt om DNA-sekvenserna på våra kromosomer.
F: Vad är det humana genomprojektet?
S: Human Genome Project (HGP) är ett projekt som tog fram en referenssekvens av det mänskliga genomet.
F: Hur många procent av sekvensen har fyllts i enligt förbättrade utkast?
S: Förbättrade utkast som tillkännagavs 2003 och 2005 fyllde i ≈92% av sekvensen.
F: Vilket är det senaste projektet som studerar hur gener kontrolleras?
S: Det senaste projektet, ENCODE, studerar hur gener kontrolleras.
F: Även om sekvensen för det mänskliga genomet har fastställts fullständigt, är den fullständigt förstådd?
S: Nej, sekvensen hos det mänskliga genomet är ännu inte helt klarlagd.
F: Vad gör icke-kodande DNA inom genomet?
S: Icke-kodande DNA i genomet gör viktiga saker som att reglera genuttryck, organisering av kromosomer och signaler som styr epigenetisk nedärvning.
Sök